① 智能医学影像
非酒精性脂肪性肝病(Non-Alcoholic Fatty Liver Diseases, NAFLD)病程的精准判断对肝病的干预治疗至关重要,是国内外重大健康问题。然而,只有少数经验丰富的病理科医生才能准确判读,制约了NAFLD病程诊断能力下沉到基层医院和偏远地区。基于NAFLD activity score(NAS)临床评分,我们拟研发出基于肝组织病理切片图像识别的多项数字化指标,精准(定量)、客观(均一化)、标准化(自动)辅助医生诊断出脂肪性肝炎、肝纤维化的潜在风险人群。目前,我们利用多示例学习(Multiple Instance Learning)人工智能方法建构出肝脂肪变性分类识别模块,其平均准确率达76%;进一步搭建的计算机底层图像处理技术的脂滴识别分析模块,可以获得高达86.6%的脂滴识别准确率(Dice Coefficient);整合后,两个肝脂肪变性识别模块平均用于每张图像的综合诊断运行时间可以短至2-3分钟。另外,使用该肝脂肪变性病理诊断模块,可以获得至少4种脂滴定量新指标:脂肪占比(T)、脂滴数量(N)、脂滴面积大小(A)与脂滴定位(P);进一步结合患者的流行病学调查数据进行相关性分析,发现指标T与指标N和S score高度相关,并且生化指标中的肝功能指标(ALT、AST)也与S score高度相关,这些结果与临床诊断经验相符;而指标A和炎症指标L score有相关性是本课题的新发现,有待后续挖掘作为新的临床诊断标志。我们拟继续使用多示例学习、目标检测与识别、注意力机制等贴合医生临床诊断要求的方法搭建完成L score与B score的诊断识别模块,开发出一套基于NAS临床评分的人工智能诊断脂肪性肝病病程精准识别系统。
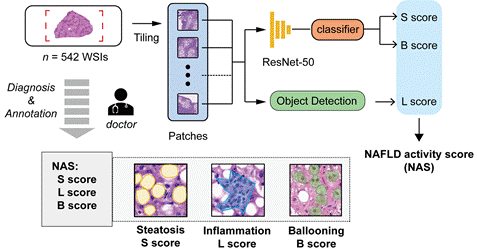
图为人工智能诊断脂肪性肝病病程精准识别系统
② 智能质谱方向
代谢物质谱检测能够全面反应人体的代谢状态。我们希望通过蛋白质组学、代谢组学等系统生物学方法,结合人工智能技术发现疾病谱图特征、新生物标记物。然而,目前主流的质谱分析方法由于仪器和软件的问题,主要依赖于人工经验,通量很低,极大制约了医疗大数据质谱检测方法应用。我们旨在建立能够快速精准的分析靶向和非靶向代谢组学数据的人工智能算法,为后续实现基于代谢组学的精准医疗,实现代谢组学与其他组学的数据整合和信息挖掘提供有力的支撑。目前主流软件在计算质谱峰面积时均会一定程度上产生错误。引起计算错误的主要原因有:质谱扫描仪器在实验过程中产生的保留时间漂移;样品本身的问题导致出现多峰或质谱峰分离差;业内软件在判断质谱峰的范围时出现误差,导致部分积分,导致峰面积偏小。传统的方法是人工识别是否出现了以上问题并手动进行调整,我们致力于设计高效算法以及开发软件来自动解决面积计算错误的问题。对于由实验仪器的误差引起的保留时间漂移问题,目前我们的算法对于单个样品的所有扫描的平均计算误差可以控制在3.5%以内,对于所有样品的平均计算误差可以控制在2.0%以内。此外,我们还结合代谢组学和人工智能方法,解析了肥厚性心肌病代谢特征以及寨卡病毒导致小头症的分子机制。
③ 智能医疗方向
一方面,开发人工智能方法建立胰岛素剂量调节辅助决策系统:
中国约50%的糖尿病患者接受胰岛素治疗,不同层次的医生对胰岛素剂量调节的掌握熟悉程度差异很大。因此,开发人工智能胰岛素剂量调节辅助决策系统将有助于医生对糖尿病患者的院内外血糖管理。我们基于中山医院病历信息系统,通过机器学习方法建立了胰岛素剂量调节模型。经过初步验证,该模型相当于主治医生及以上水平。目前在进一步优化该模型,并在医院内开展多中心研究,探讨其在糖尿病患者的血糖控制上的有效性与安全性。该系统在申请中国药监局的认证,拟开展注册临床研究。另一方面,开发人工智能方法建立糖尿病低血糖预警模型:住院糖尿病患者常出现难以被发现的夜间低血糖,因此建立低血糖预测模型对预防低血糖的发生十分重要。我们已收集中山医院住院糖尿病患者7天连续血糖监测数据,并分析低血糖发生规律与影响因素。通过机器学习方法,结合身体参数、用药情况、饮食、运动、血糖动态监测等信息,建立低血糖预警模型。
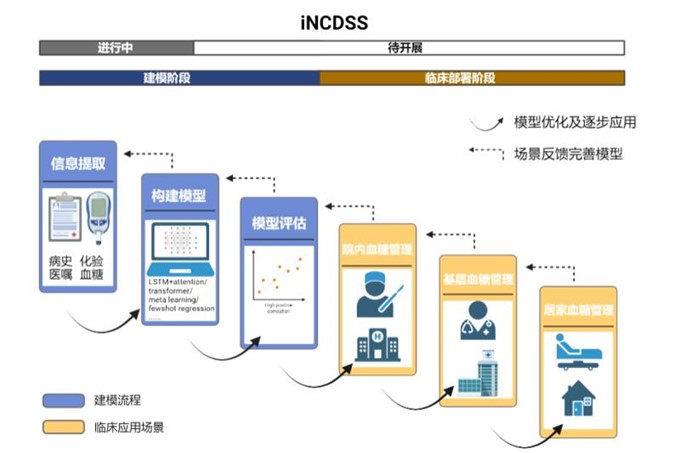
图为糖尿病智慧管理项目
④ 智能组学方向
以基因组、转录组和蛋白质组学为代表的生命组学研究通过测序、质谱等手段对生物分子的集合全体进行系统性研究,产生了大量的生命组学数据。针对生命组学数据,使用人工智能的方法进行分析、挖掘,系统研究生命体系的特征和规律,是生命科学研究的热点和前沿。我们开发了一系列方法,进行表观基因组学、转录组特别是空间转录组学、RNA结构组学,以及各种单细胞、多组学大数据的分析、整合、挖掘和功能预测。我们开发的表观基因组学单细胞数据分析的人工智能方法SCALE,结合变分自编码器和高斯混合模型进行数据降维,并通过发掘细胞特异的染色质图谱模式填补数据缺失,为研究者们解码单细胞表观遗传学提供了有力工具(见图); SCALEX方法通过将细胞映射到批次无关的广义低维空间中,从而校正单细胞测序数据中的批次效应、整合异构单细胞测序数据;StructureImpute方法将RNA序列与其对应的RNA结构信号相结合,通过自监督式训练建立了一种恢复实验缺失的RNA结构信号。另外,正在开发CARIA方法利用深度神经网络对单细胞多组学数据进行整合和信息挖掘,解析染色质开放区间与基因表达潜在的调控开关;正在开发空间转录组学的新方法SCAPE,通过整合空间信息和单细胞转录组学数据,实现位置相关的单细胞分型和聚类,并发现功能相关的单细胞区块。
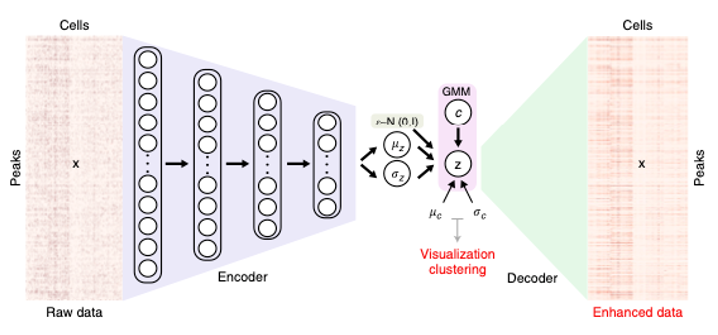
图为表观基因组学单细胞数据分析的人工智能方法SCALE